Contenu:
◉ Généralité
Le Staphylocoque figure parmi les bactéries les plus importantes en microbiologie médicale, que ce soit en termes de prévalence de virulence, de pouvoir pathogène ou de résistance aux antibiotiques.
La capacité de distinguer les staphylocoques et de différencier entre leurs différentes espèces dans une culture bactérienne est une compétence fondamentale en microbiologie.
Traditionnellement, les membres du genre Staphylococcus ont été classés en fonction de leur capacité à coaguler le plasma de lapin, se divisant ainsi en deux catégories :
- Ceux qui sont à coagulase positive, c'est-à-dire Staphylococcus aureus,
- Ceux qui sont à coagulase négative, c'est-à-dire staphylocoques à coagulase négative (CONS ou SCN).
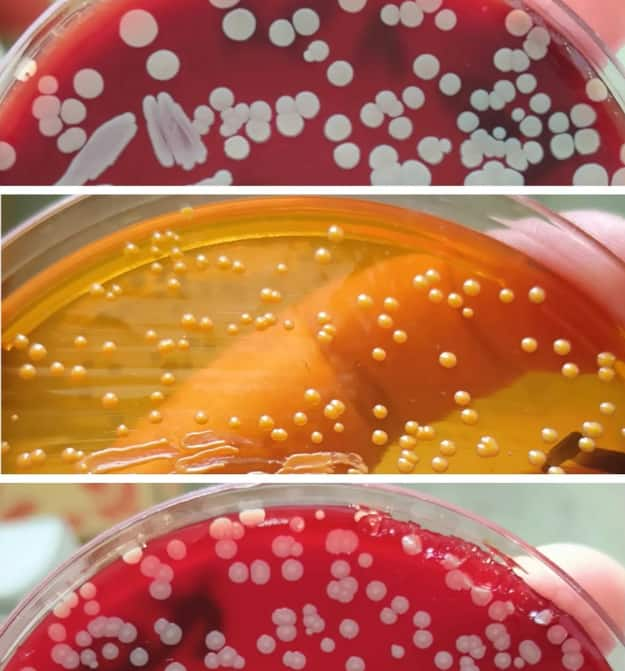
Staphylococcus aureus sur différents milieux
◉ Les étapes
L'identification des staphylocoques dans une culture bactérienne est souvent facilitée par une série de tests et un raisonnement logique. Voici les principales étapes :
- Milieux de culture: Examinez le type de milieux utilisés (sélectif, enrichi, chromogénique, etc.), la forme, le volume et la couleur des colonies.
- Coloration de Gram: Pour apprécier la forme des bactéries (cocci ou bacilles), la coloration Gram positif ou Gram négatif, ainsi que l'agencement des bactéries en amas ou en chaînette.
- Test de catalase : Effectuez ce test pour distinguer les staphylocoques (catalase+) des streptocoques et entérocoques (catalase-).
- Tests comme le test de coagulase, le test d'agglutination, et le test DNase sont principalement utilisés pour différencier S. aureus des autres staphylocoques dits à coagulase négative.
- Une identification plus complète est possible grâce à des galeries API, des automates d'identification ou par biologie moléculaire
◉ Culture et Milieux de Culture
Pendant une incubation à 35 °C sur une période de 24 à 48 heures, les staphylocoques présentent une croissance rapide sur divers milieux. Les colonies varient de 1 à 3 mm de diamètre en 24 heures et de 3 à 8 mm après 72 heures d'incubation.
Il convient de noter que des exceptions existent, telles que celles de S. aureus subsp. anaerobius, S. saccharolyticus, S. auricularis, S. equorum, S. vitulinus et S. lentus, qui ont une croissance plus lente et nécessitent généralement de 24 à 36 heures pour un développement de colonies détectable
Sur gélose au sang, Staphylococcus aureus forme des colonies typiques pigmentées, présentant une couleur variant de crème/gris/gris-blanc avec une teinte jaunâtre allant du jaune à l'orange. Ces colonies sont lisses, entières, légèrement surélevées, et démontrent un caractère hémolytique.
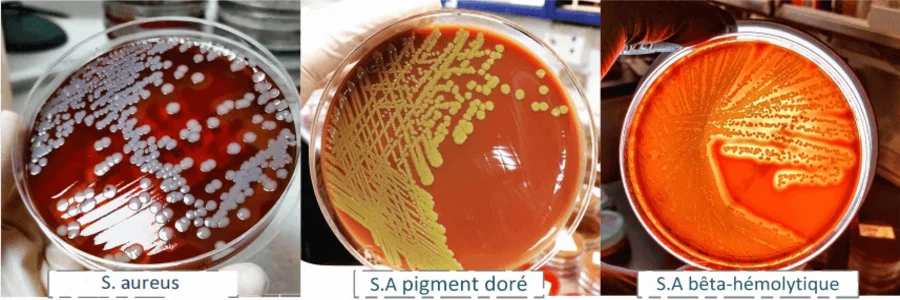
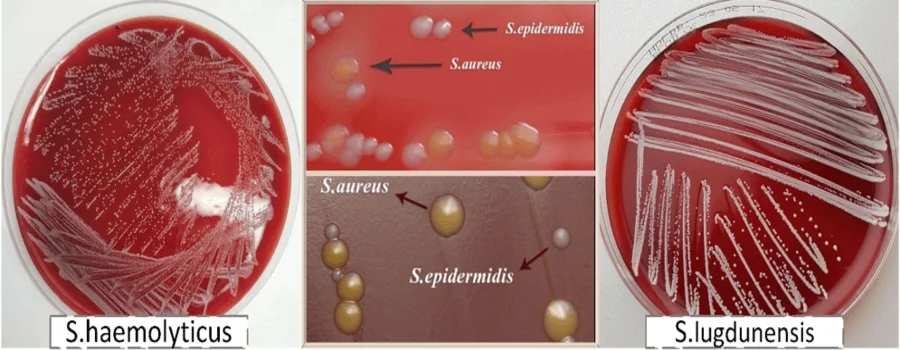
Les staphylocoques à coagulase négative (SCN), notamment S. epidermidis, présentent des colonies blanches, bien que certaines souches et espèces de SCN puissent développer des colonies avec un léger pigment crème.
Généralement, les souches SCN ne sont pas hémolytiques, bien que certaines puissent produire une petite zone de bêta-hémolyse sur la gélose au sang, comme c'est le cas pour S. haemolyticus et S. lugdunensis.
◇ Les variants formant colonies naines (VCS) de Staphylococcus sont caractérisés par des colonies ponctuelles (1/10 de la taille du type sauvage), pour la plupart non pigmentées et non hémolytiques après 24 à 72 h d'incubation
Les VCS sont souvent mélangés avec des colonies affichant le phénotype normal, donnant ainsi l'apparence d'une culture mixte (une caractéristique phénotypique partagée par plusieurs souches hétéro-résistantes de SARM).
Lors du repiquage, ils peuvent rester stables ou revenir au type sauvage (En fonction de leur auxotrophie, la croissance normale peut être rétablie si l'isolat est cultivé en présence d'hémine, de ménadione ou de thymidine et / ou une supplémentation en CO2).
☰ Figure (1):
- Flèches: Le phénotype normal est constitué de colonies grisâtres avec une teinte jaunâtre entourée d'une zone d'hémolyse
- Flèches en pointillés: le phénotype de petite colonie est caractérisé par de minuscules colonies non hémolytiques et non pigmentées
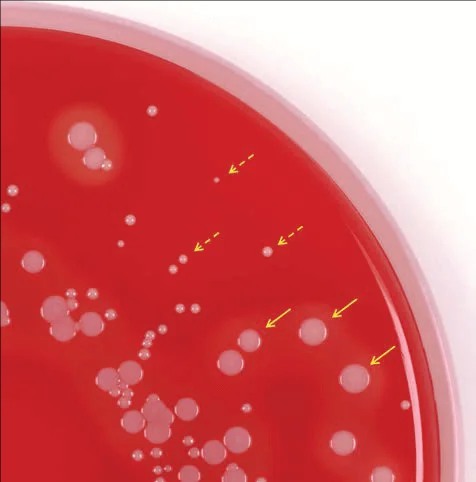
Figure (1)- Flèches en pointillés : variants formant colonies naines
☰ Comme S. aureus est fréquemment isolé dans des cultures mixtes, des milieux sélectifs et différentiels sont utilisés pour faciliter la détection de ces organismes dans le matériel clinique :
- Sur milieu de Chapman : Colonies jaunes pour le S.aureus (fermentation du mannitol)
- Milieu de Baird-Parker : surtout utilisé en bactériologie alimentaire. Le S.aureus donne des colonies noires (réduction du tellurite) avec un halo clair suivi d'une opacification tardive du halo (lipase).
- Les milieux chromogènes : fondés sur la dégradation d'un substrat en métabolite coloré spécifique d'espèce.
◉ Staphylococcus Identification du genre
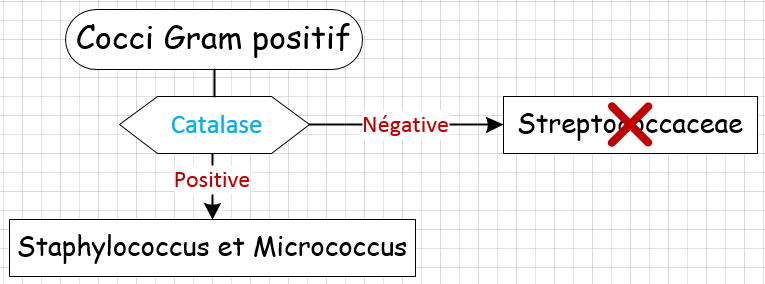
Le premier trait caractéristique qui permet de distinguer le staphylocoque est qu'il s'agit d'une cocci gram-positif (observable sous microscope après une coloration de Gram). Cependant, il est important de noter que d'autres bactéries telles que les streptocoques, les enterocoques et les microcoques partagent également cette caractéristique.
Le deuxième caractère recherché chez les staphylocoques est qu'ils sont catalase positifs. positifs. Cette caractéristique permet de distinguer les staphylocoques et microcoques qui sont catalase positifs, des entérocoques et des streptocoques qui sont catalase négatifs.
Note: La catalase est un caractère constant chez les staphylocoques, malgré quelques rares cas rapportés de S. aureus à catalase négative .
La différenciation entre le genre Staphylococcus et le genre Micrococcus n'est pas toujours évidente en raison d'une ressemblance des colonies sur gélose et le catalase positive. Cependant, il existe certains critères permettant de distinguer entre les deux genres :
| Staphylococcus | Micrococcus | |
|---|---|---|
| Oxydase | - | + |
| Bacitracine | (R) | (S) |
| Nitrofurantoine | (S) | (R) |
| Composé vibriostqtique O129 | (R) | (S) |
| Sensibilité à la lysostaphine | (S) | (R) |
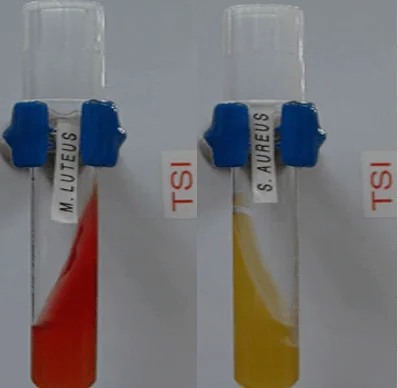
Contrairement aux staphylocoques, les microcoques sont caractérisés par l'absence de production d'acide à partir du glucose dans des conditions anaérobies (non-fermentaire).
◉ Staphylococcus Identification de l'espèce
Il existe plusieurs tests de laboratoire utilisés pour différencier les différentes espèces de staphylocoques, en particulier pour distinguer Staphylococcus aureus des autres espèces (SCN). Parmi ces tests:
◉ Test de coagulase
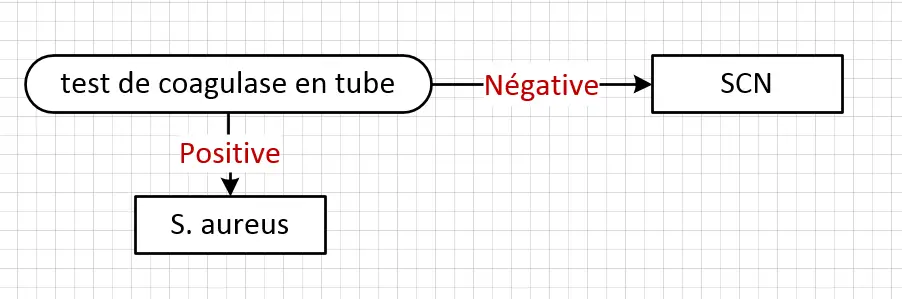
La capacité de coaguler le plasma est un critère largement utilisé en laboratoire pour différencier le Staphylococcus aureus des autres espèces. Il existe deux méthodes :
- La détection de la coagulase libre extracellulaire se fait par le test en tube, grâce à la coagulase staphylococcique qui convertit le fibrinogène en fibrine.
- La détection de la coagulase liée à la paroi cellulaire, c'est-à-dire le facteur d'agglutination (clumping factor), s'effectue par le test d'agglutination sur lame.
1- Test de coagulase en tube
- Le test de coagulase en tube implique le transfert d'une grande colonie isolée d'une gélose non inhibitrice dans 0,5 ml de plasma de lapin reconstitué.
- L'incubation du tube à 37 °C pendant 4 h est essentielle, et la formation de caillots est observée en inclinant lentement le tube à 90 ° de la verticale.
- Tout degré de coagulation est considéré comme un test positif, excluant les précipités floculants ou fibreux enregistrés comme négatifs.
- En l'absence de caillot après 4 h, le tube est relu après 18 h d'incubation à 37 °C.
- Des résultats faussement négatifs peuvent survenir pour certaines souches produisant de la staphylokinase (fibrinolysine), capables de lyser le caillot après sa formation, généralement après une incubation prolongée.
Note: Lors de l'utilisation d'un plasma provenant d'un tube citraté pour le test de coagulase en tube, il est important de noter que des organismes tels que Enterococcus faecalis, Pseudomonas, Serratia marcescens, et certaines souches de Streptococcus, qui utilisent le citrate, peuvent consommer ce dernier, entraînant un résultat faussement positif.
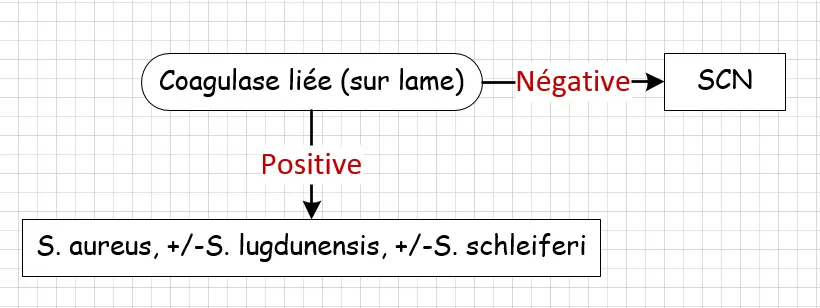
1- Test de coagulase sur lame
- La coagulase liée (facteur d'agglutination) est détectée par un test d'agglutination sur lame, impliquant l'émulsion d'une suspension de l'organisme sur une lame avec une goutte de plasma de lapin, provoquant l'agglutination en présence de coagulase liée.
- Pour une interprétation correcte, un contrôle utilisant une solution saline à la place du plasma est nécessaire pour vérifier l'auto-agglutination.
- Parmi les SCN, S. lugdunensis et S. schleiferi peuvent également tester positif pour la coagulase liée.
- Moins sensible et moins spécifique que la coagulation en tube,

Pour plus d'informations sur le test de coagulase, veuillez consulter notre article : "Test de coagulase"
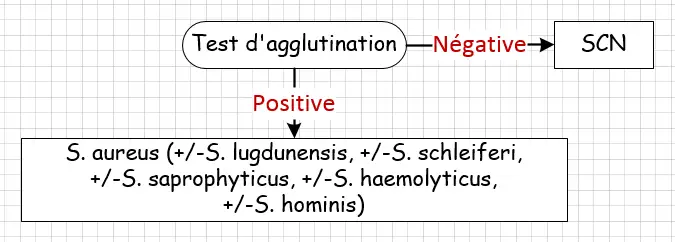
◉ Test d'agglutination
Afin de surmonter les limitations telles que la faible sensibilité et spécificité du test d'agglutination sur lame classique, ainsi que le long temps d'incubation du test de coagulase en tube, des alternatives plus rapides ont été développées. Parmi celles-ci, on compte les tests rapides au latex et d'hémagglutination, qui permettent une identification présomptive rapide de Staphylococcus aureus.
Le test repose sur l'utilisation d'anticorps spécifiques dirigés contre des antigènes de surface de Staphylococcus aureus. Lorsque ces anticorps réagissent avec les antigènes correspondants sur les cellules bactériennes, cela entraîne la formation d'agrégats, provoquant une agglutination visible.
Les tests de troisième génération comprennent l'utilisation d'anticorps monoclonaux qui reconnaissent les sérotypes de polysaccharides capsulaires les plus couramment rencontrés sur le plan clinique (1, 5, 6, 8, 10).
Parmi ces tests, on peut citer Pastorex Staph-Plus (Bio-Rad), Slidex Staph Plus (bioMérieux), Staphaurex Plus et Staphytect Plus (Oxoid).
Note: Cette technique doit être interprétée prudemment en raison de la possibilité de résultats faussement positifs ou faussement négatifs.
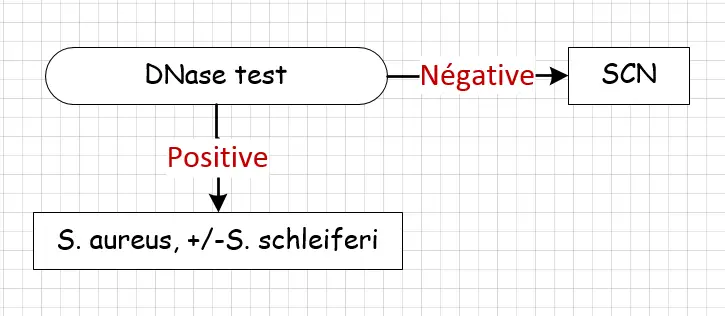
◉ DNase test
- S. aureus et S. schleiferi possèdent des enzymes nommées désoxyribonucléases capables de dégrader l'ADN.
- La gélose à l'acide désoxyribonucléique (A.D.N.) est un milieu conçu pour mettre en évidence l'activité de la désoxyribonucléase des bactéries.
- La zone d'hydrolyse est révélée par l'ajout d'acide chlorhydrique à la surface du milieu, provoquant la précipitation de l'ADN et l'opacification du milieu.
- La présence d'un halo clair autour de la culture indique que l'ADN a été hydrolysé.
Pour plus d'informations sur le test de DNase, veuillez consulter notre article : "Test Désoxyribonucléase (DNase)"
◉ La résistance à la novobiocine
La résistance à la novobiocine est inhérente à S. saprophyticus, mais elle est rare chez d'autres espèces de staphylocoques cliniquement importantes, et cette résistance est donc utilisée pour distinguer S. saprophyticus des autres staphylocoques.
- Un test de diffusion sur gélose pour évaluer la sensibilité à la novobiocine peut être effectué à l'aide d'un disque de novobiocine de 5 µg sur gélose Mueller-Hinton.
- Avec une suspension d'inoculum équivalente en turbidité à un standard d'opacité McFarland de 0,5 et une incubation à 35 à 37°C pendant une nuit ou jusqu'à 24 heures, la résistance à la novobiocine est indiquée par un diamètre de zone d'inhibition ≤ 16 mm.
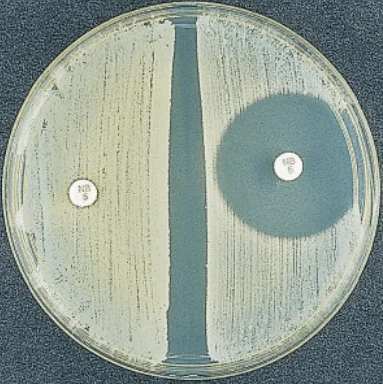
◉ Test ornithine décarboxylase
- Test ornithine décarboxylase pour l'identification de Staphylococcus lugdunensis.
- Contrairement à la plupart des autres espèces staphylocoques à coagulase négative, S. lugdunensis est ornithine décarboxylase positive.
- Le milieu décarboxylase contenant 1% d'ornithine est inoculé et incubé pendant une nuit.
- Étant donné que certaines souches de S. epidermidis peuvent également être positives à 24 h, l'échantillon doit être examiné à 8 h, moment auquel S. lugdunensis est positif mais S. epidermidis est toujours négatif.

S. lugdunensis (à droite) est positif. S. saprophyticus (à gauche) est négatif
◉ L'identification complète
Pour une identification complète des staphylocoques, plusieurs méthodes peuvent être utilisées, notamment:
◉ Identification biochimique
L'identification biochimique complète des staphylocoques implique l'utilisation de tests qui évaluent différentes caractéristiques métaboliques et biochimiques des bactéries.
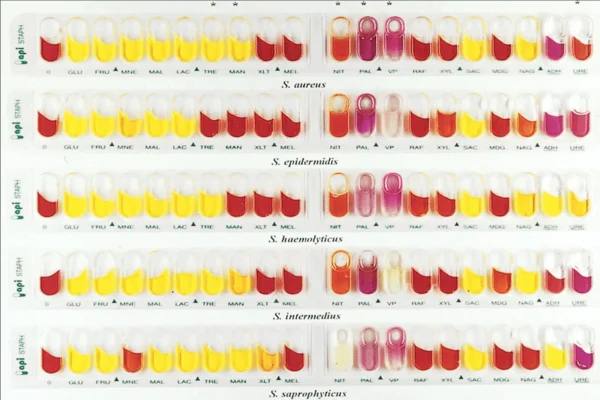
1- La galerie biochimique d'identification
les galeries biochimiques d'identification telles que API Staph®, ID 32 Staph®, et RAPIDEC Staph® sont des systèmes de tests standardisés utilisés pour caractériser et identifier les staphylocoques.
Les résultats de ces tests sont interprétés en fonction de profils métaboliques spécifiques, facilitant ainsi la classification des staphylocoques en différentes espèces ou souches.
Après incubation de 24H vous allez lire votre API en fonction des variations des couleurs. Télécharger le manuel API staph pdf
◉ Identification basées sur les acides nucléiques
L'identification des staphylocoques par acides nucléiques utilise des techniques de biologie moléculaire basées sur l'analyse de l'ADN.
- L'extraction des acides nucléiques des staphylocoques Gram-positifs peut être complexe et nécessite des conditions spéciales de lyse cellulaire, impliquant des enzymes telles que la lysostaphine, la lysozyme, la protéinase K et l'achromopeptidase.
- Des tests d'amplification spécifique ciblant des fragments des gènes universels d'ARNr 16S et 23S sont utilisés pour l'identification des staphylocoques.
- D'autres cibles ADN universelles comprennent les gènes tuf, gyrA, sodA, gap, et HSP60 / GroE.
- Pour l'identification spécifique de S. aureus, le gène nuc (codant pour la nucléase thermostable) est une cible populaire et hautement spécifique, avec des méthodes de PCR spécifiques pour ce gène.
- D'autres cibles spécifiques utilisées pour l'identification de S. aureus incluent les gènes clfA, coa, sodM, femA, femB, et l'élément d'insertion staphylococcique 431 (uniquement pour le SARM).
Note: Il est important de noter que des erreurs d'identification peuvent se produire, notamment en raison de souches de S. aureus fem négatives et de CoNS ayant des gènes liés à femA.
◉ Sensibilité aux antibiotiques et traitement des staphylocoques
Les anti-staphylococciques sont présents dans toutes les familles d'antibiotiques, à l'exception des polymyxines et des imidazolés. Les classes d'anti-staphylococciques les plus couramment utilisées comprennent :
- Les bêtalactamines, avec l'oxacilline du groupe des pénicillines M.
- Les glycopeptides, tels que la vancomycine ou la teicoplanine en cas de résistance ou d'intolérance à la première classe.
- Les aminosides, notamment la gentamicine, qui est souvent utilisée en association avec l'une des deux classes précédentes pour obtenir une bactéricidie rapide.
- Les fluoroquinolones.
D'autres antibiotiques sont également employés, tels que les streptogramines (pristinamycine), les macrolides, la clindamycine, le cotrimoxazole, la rifampicine, l'acide fusidique et la fosfomycine. Ces trois dernières molécules sont souvent administrées en association en raison des fréquences élevées de mutations.
◈ Pour obtenir davantage d'informations sur la réalisation d'un antibiogramme, veuillez consulter notre article: "Antibiogramme"
◈ Pour des détails spécifiques sur l'antibiogramme des staphylocoques, consultez notre article: "Antibiogramme de Staphylocoque"
◉ Conclusion
En conclusion, l'identification précise des staphylocoques revêt une importance capitale dans le domaine de la microbiologie médicale. Les avancées constantes dans les techniques d'analyse, des tests biochimiques traditionnels aux méthodes de biologie moléculaire innovantes, ont considérablement amélioré notre capacité à caractériser ces bactéries pathogènes.
Reference
- - NCBI/GenBank:Taxonomy-Browser
- - American society for microbiology
- - Biorad : PASTOREX™ STAPH-PLUS manual
- - Staphaurex Plus manual
- - Planche : Diagnostic bactériologique staph , DR.Hamzaoui lina
- - Kateete et al. Annals of Clinical Microbiology and Antimicrobials, Identification of Staphylococcus aureus: DNase and Mannitol salt agar
- - T. Rasamiravaka et al, Human Plasma Tube Coagulase Test for Identification of Staphylococcus aureus : Can we routinely use it ?
- - UK Standards for Microbiology Investigations
- - tcs bioscience : RABBIT COAGULASE PLASMA
- - Journal of Laboratory Physicians : Mannitol‑fermenting and Tube Coagulase‑negative Staphylococcal Isolates:
- - HAL : Restoring catalase activity in Staphylococcus aureus subsp. anaerobius leads to loss of pathogenicity for lambs
- - Manual of clinical microbiology 11ed
- - Textbook of Microbiology 4ed